In questo articolo concluderemo il nostro excursus sui meccanismi di resistenza tramite i quali i microrganismi mettono si difendono dall’azione terapeutica degli antibiotici.
Fino ad adesso abbiamo approfondito le strategie di modifica del target antimicrobico e dei meccanismi di assorbimento/estrusione dell’antibiotico dalla cellula batterica.
In questa sede, invece, l’attenzione verrà posta sugli ultimi due meccanismi di resistenza batterica:
- cambiamenti e/o bypass dei siti bersaglio;
- cambiamenti globali in importanti vie metaboliche attraverso la modulazione delle reti di regolazione.
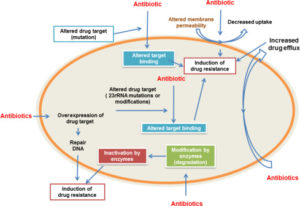
MODIFICHE E/O BYPASS DEI SITI BERSAGLIO
Una delle strategie che i batteri mettono in atto per sviluppare resistenza all’azione di un antibiotico è quella di modificare, in modi diversi, il suo sito bersaglio.
In particolare, i batteri sono in grado sia di proteggere il sito di legame, evitando che la molecola antibiotica possa raggiungerlo, sia di modificarlo e, quindi, di diminuire significativamente l’affinità dell’antibiotico al sito di legame stesso.
Questo tipo di meccanismi di resistenza si possono riassumere in due punti:
- Protezione del sito di legame;
- Modifiche del sito di legame.
Protezione del sito di legame (bersaglio)
Alcuni dei determinanti genetici che codificano per proteine che mediano la protezione del bersaglio sono stati trovati nel cromosoma batterico.
Esempi di farmaci interessati da questo meccanismo sono le tetracicline (Tet [M] e Tet [O]), i fluorochinoloni (Qnr) e l’acido fusidico (FusB e FusC).
Per quanto riguarda, in particolare, la resistenza alle tetracicline Tet (M) è stato inizialmente descritto in Streptococcus spp. e Tet (O) in Campylobacter jejuni, ma ora sono entrambi ampiamente distribuiti tra diverse specie batteriche.
Queste proteine appartengono alla superfamiglia dei fattori di traduzione delle GTPasi e agiscono come omologhe dei fattori di allungamento (EF-G e EF-Tu) utilizzati nella sintesi proteica. TetO e TetM interagiscono con il ribosoma e rimuovono la tetraciclina dal suo sito di legame in modo GTP-dipendente.
TetM rimuove e rilascia direttamente la tetraciclina dal ribosoma mediante un’interazione tra il dominio IV dell’rRNA 16S e il sito di legame della tetraciclina. Inoltre, questa interazione altera la conformazione ribosomiale, impedendo il re-binding dell’antibiotico.
Allo stesso modo, è stato anche dimostrato che TetO compete con la tetraciclina per lo stesso spazio ribosomiale e altera la geometria del sito di legame dell’antibiotico. Cos’ facendo, sposta la molecola dal ribosoma e consentendo la ripresa della sintesi proteica.
Modifiche al sito di legame
L’introduzione di modifiche al sito bersaglio è uno dei meccanismi più comuni di resistenza agli antibiotici nei patogeni batterici ed interessano quasi tutte le famiglie di composti antimicrobici.
Questi cambiamenti del target possono consistere in:
- mutazioni puntiformi nei geni che codificano il sito target;
- alterazioni enzimatiche del sito di legame (ad esempio aggiunta di gruppi metilici);
- sostituzione o bypass del target originale.
Come accennato, indipendentemente dal tipo di cambiamento, l’effetto finale è sempre lo stesso: una diminuzione dell’affinità dell’antibiotico per il sito target.
Mutazioni puntiformi nei geni che codificano il sito target
Uno degli esempi più classici di resistenza mutazionale è lo sviluppo della resistenza alla rifampicina (RIF).
Questo antibiotico blocca la trascrizione batterica inibendo la RNA polimerasi DNA-dipendente. La tasca di legame della rifampicina è una struttura altamente conservata situata nella subunità β della RNA polimerasi e, dopo il legame, la molecola antibiotica interrompe la trascrizione bloccando direttamente il percorso dell’RNA nascente.
È stato dimostrato che la resistenza a RIF si verifica mediante mutazioni puntiformi a singolo passaggio che determinano sostituzioni di amminoacidi nel gene rpoB e sono stati segnalati molti cambiamenti genetici differenti. Queste mutazioni provocano una diminuzione dell’affinità del farmaco per il suo bersaglio, di solito risparmiano l’attività catalitica della polimerasi, consentendo la trascrizione di continuare.
Alterazioni enzimatiche del sito di legame
Uno degli esempi meglio caratterizzati di resistenza mediata dalla modifica enzimatica del sito bersaglio è la metilazione del ribosoma catalizzata da un enzima codificato dai geni erm (metilazione ribosomiale dell’eritromicina), che si traduce in resistenza ai macrolidi. Questi enzimi sono in grado di mono- o di-metilare un residuo di adenina in posizione A2058 del dominio V della subunità ribosomiale 50S.
A causa di questo cambiamento biochimico, il legame della molecola antimicrobica al suo bersaglio è compromesso. È importante sottolineare che, poiché macrolidi, lincosamidi e antibiotici streptogramina B hanno siti di legame sovrapposti, l’espressione dei geni erm conferisce resistenza crociata a diversi antibiotici.
Sostituzione o bypass del target originale
Utilizzando questa strategia, i batteri sono in grado di sviluppare nuovi bersagli che svolgono funzioni biochimiche simili al bersaglio originale, ma non sono inibiti dalla molecola antimicrobica.
Gli esempi clinici più rilevanti includono la resistenza alla meticillina in S. aureus, dovuta all’acquisizione di una Penicillin Binding Protein esogena (PBP2a), e la resistenza alla vancomicina negli enterococchi attraverso modifiche della struttura del peptidoglicano mediata dai cluster del gene van.
Infine, un altro modo per evitare l’azione antimicrobica è quello di “bypassare” la via metabolica che inibiscono producendo in eccesso il bersaglio antibiotico. Un esempio rilevante di questo meccanismo è la resistenza al trimetoprim-sulfametossazolo (TMP-SMX).
RESISTENZA DOVUTA AD ADATTAMENTI CELLULARI GLOBALI
In anni di evoluzione, i batteri hanno sviluppato meccanismi sofisticati per far fronte a stress e pressioni ambientali per sopravvivere agli ambienti più ostili, compreso il corpo umano.
I batteri devono competere per i nutrienti ed evitare l’attacco di molecole prodotte da altri organismi rivali per avere il “sopravvento”. All’interno di un particolare ospite, gli organismi batterici sono costantemente attaccati dal suo sistema immunitario e, per stabilirsi in particolari nicchie biologiche, è fondamentale che si adattino e facciano fronte a queste situazioni di stress.
Pertanto, i patogeni batterici hanno escogitato meccanismi molto complessi per evitare l’interruzione del processo cellulare fondamentale come la sintesi della parete cellulare e l’omeostasi della membrana.
Lo sviluppo di resistenza alla daptomicina (DAP) e alla vancomicina (basso livello in S. aureus) sono gli esempi clinicamente più rilevanti di fenotipi di resistenza che sono il risultato di una risposta adattativa cellulare globale all’attacco antibatterico.







